- 09/28
- 2022
-
QQ扫一扫

-
Vision小助手
(CMVU)
荧光显微镜是阐明蛋白质和其他分子的细胞功能的最强大工具之一。在许多情况下,可以通过比较荧光标记分子的分布,推断出特定细胞内结构或分子复合物的关联。尽管荧光显微镜是生物医学研究中最常见的应用,但很少量化评估两个荧光分子“共定位”。
荧光共定位分析适合确定两个分子是否相关联:例如,标记的分子是否与内体、线粒体或微管相关联,或两种蛋白是否与具有相同的亚结构或与相同的质膜结构相关联。共定位受到分辨率的限制;荧光的重叠并不一定证明两个探针在同一细胞结构中。然而,观察到两个探针在整个细胞的多个结构中的反复重合,说明两者很可能具有相同结构域。
共定位可以被认为由两个部分组成:一、共现,两个探针的简单空间重叠;二、相关性,此时两个探针不仅彼此重叠,而且在结构内和结构之间按比例分布。例如,内体中被GFP标记的Rab蛋白与其内部的蛋白分子应该是共现的关系,因此它们的荧光表达量并不一定成比例。如果被标记的分子在空间上是相关的,那么这两个探针彼此会按一定比例重叠分布。
1. 共定位分析前的准备工作
在共定位分析方法介绍前,首先需要注意样品制备和成像的准备工作,好的图像能够避免共定位分析错误。例如,不能串色(否则共定位会出现假阳性);使用亮度或特异性好的染料,提高成像信噪比(SNR);如果是双相机还需要考虑相机的校准对齐;成像中可能发生的色差问题、漂移问题都需要预防或者后期图像处理矫正。
2. 共定位定性分析
共定位定性分析是通过视觉来判断共定位。例如常见的两个染料红色和绿色,信号叠加的部分会变为黄色。因为荧光成像的颜色为伪彩,可以轻松变换为其他颜色,现在也有青色和品红的搭配,青色和品红叠加为白色。
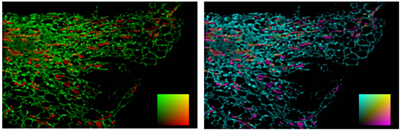
图1 两通道图像使用不同伪彩搭配展示
这种方法优点在于直观,但是并非所有红绿叠加一定会为黄色,因为只有两种信号的强度相似时才能获得黄色。此外红绿色盲人群将无法使用这种方法。共定位定性分析技术的缺点很明显,因此需要一种更客观的分析方法来量化共定位结果。
3. 共定位定量分析
3.1. 共现:Mander系数
Mander 介绍了一种在考虑像素强度的同时确定两个图像的重叠的方法,我们称之为共现。先介绍两个系数“共定位系数M1和M2”:
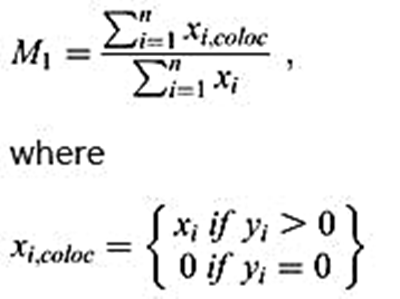
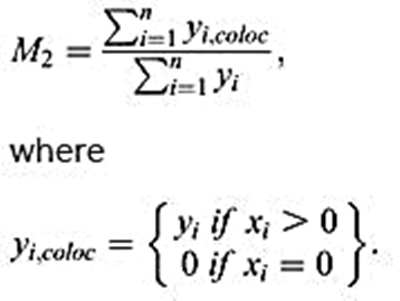
这里,xi和yi分别指通道1和通道2的第i个高于背景阈值的像素值,每个图像中共n个像素被分析。仅当对应的yi和xi也分别高于阈值时,xi,coloc和yi,coloc才可以具有非零值。因此,M1可以定义为通道1对应通道2的共现分数,也称共定位系数MCC。M2定义类似。
这些系数避免了与信号的绝对强度有关的问题,因为它们是相对于总像素强度进行归一化的。我们还能得到每个通道重叠程度的信息。但是MCC很容易受到背景信号的影响,因此很关键的步骤就是要如何设定一个阈值来去除背景信号的干扰。
消除背景最简单的方法是从图像的像素强度中减去一个全局阈值。然而,对于许多图像,判断背景阈值具有挑战性。更重要的是,对于荧光强度接近背景强度的图像中,MCC 对阈值的选择非常敏感。阈值的轻微变动就会导致 MCC 发生大的变动。因此需要一个能够自动判断阈值的方法非常重要,后续我们会再提到具体方法(见3.4)。
此外,Mander 提出了一个整体重叠系数 (MOC):
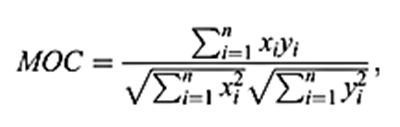
MOC仅以强度加权的方式表示两个结构在空间上重叠的程度。MOC对更亮的像素赋予更大的重要性,而对接近零或阈值的值赋予更少的权重(图2)。
MOC对信噪比 (SNR) 的差异相对不敏感,图2A说明了这种现象。可以看出,SNR降低(从19到8)仅导致MOC值发生 3% 的相对较小的变化。虽然对波动的SNR具有鲁棒性,但准确的MOC值取决于有效去除不需要的信号。因为不需要的信号会增加像素强度,应该始终小心地从图像中减去它们。图2B说明了这种效果。在左侧,显示的假设图像仅包含感兴趣的信号和伴随的噪声,其MOC值为0.691。添加了相对较暗的散焦光(中间图像),显示为实际亮度的两倍,以创建右侧显示的图像。结果,与原始图像相比,MOC值增加了 20%(0.691对0.842),从而给人一种更高共现的假象。

图2 Mander的重叠系数对信噪比不敏感,但对离焦信号敏感。(A) 降低信噪比 (SNR) 对 Mander 重叠系数 (MOC) 值的影响相对较小。在此示例中,SNR 降低 60% 只会导致图像的 MOC 降低 3%(从左到右)。在计算 MOC 值期间,当对许多像素求和时,由于噪声导致的像素强度的随机波动在很大程度上被抵消了。(B) 失焦信号影响的说明。此处,原始图像(左)显示的 MOC 值为 0.69。添加具有低空间频率的相对低强度背景信号(中图)后,即使在阈值已被正确调整,所得图像(右)显示 MOC 值增加 >20%。
因此MOC一方面对信噪比SNR不敏感,另一方面要注意去除背景信号和其他杂散光和非特异性信号的干扰。此外还要注意的是,整体重叠系数MOC受两通道信号比例的影响很大,假设信号强度等于染料分子(这不一定是正确的),只有在红绿像素数量大致相等的情况下才会使用MOC (即Nred÷Ngreen像素~1)。

图3 红绿像素数差异大导致MOC(图中Overlap coeff. (R))偏低
MOC作为共定位系数,相对MCC来说具有很多缺点,无法像MCC反映出两通道共定位的相互关系,例如图3中,此时重叠系数MOC偏低只有0.444,但MCC可以发现M2的值高达0.989,同时M1只有0.199,说明此时有部分通道1的信号并没有与通道2的信号重叠,这在MOC中无法体现。而且MOC容易收到非特异型信号的影响,导致在共定位分析过程中,容易出现假阳性结果;同时对样品信号的变化感知不明显,MOC的结果具有很大的迷惑性。
一般来说,Mander系数可用于评估结构或分子在特定位置或细胞器中的存在程度。例如,它已被用于量化感兴趣的分子与线粒体(Bravo等人,2011年;Seibler等人,2011年)、质膜(Yeung等人,2008年;Spira等人,2012年)或内质网 (Horner et al., 2011; Arruda et al., 2014)的共现。
3.2. 散点图
荧光共定位研究的结果也可以在散点图中以图形方式表示,XY轴分别代表两个颜色或通道信号强度,其中一种颜色的强度针对每个像素的第二种颜色的强度绘制,类似于为流式细胞术。在按比例分布的条件下,例如对图4A中所示的图像绘制强度散点图,散点图的点围绕一条直线分布,说明两个荧光信号之间具有相关性,其斜率反映了两个颜色的荧光比(图4D)。相反,图4E中所示的图像中缺乏共定位关系,散点图的点分布到两个独立的部分,每部分显示一个探针的不同信号水平,相对另一个探针的信号在该部分很少或没有信号(图4H)。
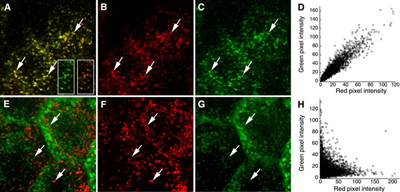
图4 内吞探针的共定位分析。A:与结合Texas Red(B)和Alexa 488 (C)的转铁蛋白孵育后,Madin Darby犬肾细胞(MDCK)图像的最大投影。箭头表示包含两种探针的核内体。D: a中两个细胞的红色和绿色像素强度散点图。E: MDCK细胞在Texas red -dextran (F)和Alexa 488-transferrin (G)孵育后图像的最大投影。箭头表示含有Texas red -dextran的溶酶体。H:图E中红色和绿色像素强度散点图。
3.3. 相关性:Pearson系数
Mander的重叠系数表示两个图像之间共现的程度,但基于相关性的共定位分析是对共定位的不同解释:如果两个成像目标在功能上相关,那么它们在同一区域的任何地方的丰度(强度)也都是可以预测的。换言之,相关性测量的是一幅图像中的信号强度与另一幅图像中的相应值之间的关系,而不是信号共现的程度。
Pearson相关系数(PCC)是衡量这种关系的可预测性的常用指标。在更数学的术语中,PCC可以被认为是两个图像之间的协方差,通过它们的标准偏差的乘积进行归一化:
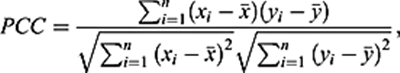
其中xi和图片分别表示的通道1图像中的第i个像素强度和平均强度(去除阈值后)。同样,yi和图片是对应的通道2图像的第i个像素强度和平均值。每个图像中共n个像素被分析。PCC值的范围从-1到1,代表荧光强度反向相关和荧光强度完全线性相关。接近0的值则反映了彼此不相关。因为从每个像素的强度值中减去平均强度,所以PCC与荧光强度和背景无关。因此,PCC可以在双色图像中测量,无需任何形式的预处理,使其既简单又相对安全,不受用户偏见的影响。
虽然PCC极值的含义通常很清楚,但中间值更难解释,除非在比较研究中使用。只要有可能,应通过阳性和阴性对照证明共定位分析的可靠性。
默认情况下分析软件会测量整个图像的PCC。然而,PCC通常应该针对单个细胞进行测量,这可以通过在图像上手绘“感兴趣区域”(ROI)来完成。这是由于PCC值依赖于简单的线性关系,如果在分析区域内有非特异性表达,或者细胞的转染蛋白表达差异等情况发生,PCC的值将偏低,因此无法准确代表相关程度。
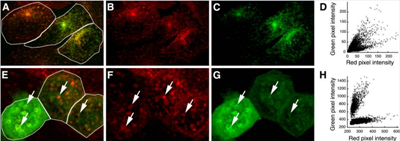
图5 PCC测量中ROI的重要性
例如上图5中,A-C由于三个细胞中本身表达的受体不同,导致与之结合的蛋白的转化效率不同,在右侧散点图D中可以看见分成了不同的三个部分。每种的红绿荧光比例不同,虽然单个细胞的PCC分别为0.88、0.85和0.89,但是整个图像的PCC降低到0.66。此外由于转染蛋白的表达在细胞之间可能有很大差异,在E-G中,左侧细胞内绿色荧光蛋白表达效率更高,与右侧细胞相比在散点图H中明显分为两个部分,虽然单个细胞的PCC分别为0.69、0.56 和 0.57,但整个图像的PCC测量值仅为0.07。
细胞周围的背景信号虽然看起来不明显,但如果将其包含在测量区域中,由于这些空白区域包含红色和绿色信号都显著低于其平均强度,而PCC的公式中单项是与平均值之间的绝对差异,因此可以人为地夸大 PCC 值。
上面的例子表明,仔细勾勒出两个探针可能分布的区域对于准确测量PCC至关重要。甚至在细胞内部也会存在不相关的区域,例如,如果分析囊泡时,此时圈选了细胞核区域,由于囊泡并不存在在细胞核内,这类似上述选择了背景区域一样,人为夸大了PCC的值。
排除不相关像素的另一种方法是将分析限制在强度高于阈值的像素。该方法一般是通过选定图像中的一些区域,认为是“背景”区域,在散点图上反映为落在靠近坐标轴的象限内,那么落在此强度范围内的像素将从分析中省略。
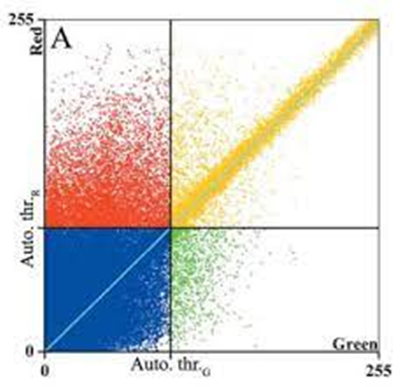
图6 通过阈值可以分割散点图为四个象限,象限交界的分割线是设定的每个通道的背景阈值
这种方法比在原始图像上手动勾勒ROI的过程要简单得多。然而,扣除的低强度像素可能包括无相关性的区域,因为无相关性的区域可能强度较低,在分析之前就被背景阈值排除了。在很多实验中无相关性也是实验的重要结果之一,因此保留这些区域具有重要的现实意义。
PCC方法的另一个潜在弱点在于PCC只适合测量单一线性关系,它在更复杂的情况下容易出现不准确的结果。例如下图7A-C中标记的两种目的蛋白位于不同的胞内结构,由于转铁蛋白的浓度含量在不同位置表达变化,无法用简单的线性模型解释,导致整体PCC值低于实际共定位程度(整体PCC为0.66)。
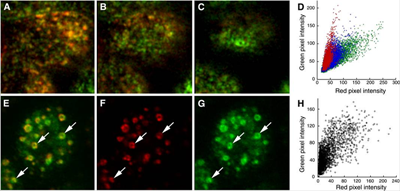
图7 没有简单线性关系的共定位。A:与Texas Red标记的转铁蛋白(红色)和Oregon Green标记的IgA(绿色)一起孵育的 MDCK 细胞内中间平面。B:与A相同,但焦面比A高1.2μm。C:与A相同,但焦面高出2.4μm。D :从A(绿色)、B(蓝色)或C(红色)中所示的焦平面收集的根尖的红色和绿色像素强度的散点图。E: MDCK 细胞免疫标记EEA1(F),和表达GFP-RhoB(G)的单个图像平面。箭头表示被两种探针共同标记的胞内结构 。H:E中显示的红色和绿色像素强度的散点图。
即使在同一个胞内结构中,由于其并不一定具有简单的线性比例关系,那么 PCC 的结果就会偏低,例如在图7 E-G中可以看见两种蛋白GFP-RhoB(绿色)和EEA1(红色)的重叠度很高,但是通过其散点图H广泛的分布范围说明其 PCC 相对较低(0.73)。
3.4. 背景阈值
通过Mander和Pearson共定位系数的介绍,我们可以注意到,排除背景阈值对两者的结果分析都有一定作用。虽然MOC对信噪比的容忍度很高,但是背景升高会直接造成MOC的虚高,对MCC来说也是如此。PCC对背景信号不敏感,但是通过对背景信号的减除可以排除非相关信号的干扰。所以有一个好的背景阈值判断方法,能够更准确的进行共定位分析。
一般来说,背景的准确估计是通过对阈值图像的视觉检查来主观评估的。例如上述图6通过选定图像中的一些区域,认为是“背景”区域。这种方法非常容易实现,但是其缺点也是不言而喻的:主观的框选很容易造成阈值过高或者过低,对于共定位系数和实验分析结果都有不小的偏差影响。
为了确定相关分析的最佳阈值,Costes等人提出了一种基于跨阈值范围计算PCC值来分割图像的方法(Costes等人,2004)。在这种方法中,提出了连续降低两幅图像的阈值,并为高于和低于这些阈值的像素计算PCC值。当PCC值接近零时,认为此时的阈值已对来自背景的信号进行了适当的分割。
无论用于选择阈值的标准如何,没有完美解决图像背景阈值分割的方法:在许多情况下,背景水平在空间上会发生变化,因此没有一个固定阈值适合整个图像。背景的空间变化可能是由照明或检测的空间不均匀性或离焦荧光引起的,即使在共焦荧光图像中也可能会出现这种情况。
3.5. Pearson相关系数与Mander共定位系数的比较
Pearson和Mander系数的应用场景不同:对于PCC来说荧光的强度相关性变化是其测量的主要目标,而Mander系数中主要判断的是荧光分子的重叠程度。Mander的MOC相比 MCC更让人迷惑一些。MOC本身用来作为PCC的互补解释,但是其对信号变化的不敏感,而对于背景信号的过于敏感,并且在很多共定位不明显的情况下MOC都具有很高的数值,导致结果难以解释,因此很多人认为MOC不适合用来测量共定位。更多推荐使用PCC和MCC来互补分析共定位。
PCC和MCC代表了生物医学研究中使用的两个主要共定位指标。严格来说,谁都不比谁优越;两者都有优点和缺点,需要根据具体情况选择使用。
MCC最明显的优势是它比PCC更直观。前述对于非简单线性相关的数据,PCC不太适合,但MCC更有用。通常,探针未按比例共同分布的情况将产生难以解释的模棱两可的中间PCC值。这些中间值可能表示数据与PCC的基础模型之间的不匹配,而不是表示部分共定位。这种情况下MCC将被认为是更好的共定位指标,因为它与信号比例无关。例如图7E中MDCK细胞中标记的EEA1(F)和GFP-RhoB(G)的共定位分析问题。
MCC另一个优势是它提供了两个相互比较的比值公式。例如如果A与B百分百重叠,但是B与A只有部分重叠,还存在于其他结构中,此时很可能获得一个PCC值很低的结果,但是通过MCC的两个比值公式就能很好的解释这种情况。如图3。
MCC的主要难点在于如何可靠的将背景与信号分开。上述的Costs方法也不能完全解决问题。面对不同的情况将需要不同的方法来解决。对于背景阈值很难判断的图像,PCC分析可能仍然是首选方法,因为它不需要任何的图像预处理。
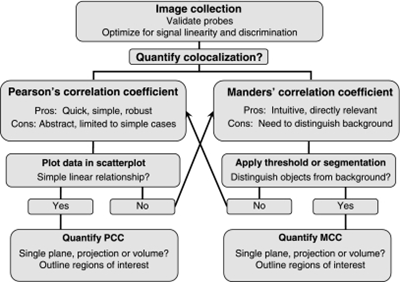
图8 一般共定位分析工作流程示意图
因此当使用共定位分析生物学问题的通用步骤应该是,研究者首先需要验证用于标记分子定位的荧光探针。图像采集系统必须进行优化,以检测最弱的信号的同时不能导致亮度高或表达高的信号发生过曝。应优化系统的光路,避免两个荧光信号之间发生串色。共定位可以通过Pearson相关系数(PCC)或Mander相关系数(MCC)进行量化。PCC分析的第一步是直观地评估像素强度数据的散点图,以评估数据是否遵循简单的线性关系。在满足这一条件的情况下,PCC可以对单个细胞或指定ROI中进行量化分析。如果数据不能遵循简单的线性关系,MCC可能是量化共定位的首选方法。MCC分析的一个初步步骤是确定背景阈值。这样,MCC可以在单个图像或指定ROI中进行量化。
4. 共定位研究中的显著性检验
共定位分析中经常被忽视的一个是需要确定在两个图像之间观察到的共现是否主要是由于随机引起的,也就是是否具有显著差异。此外对于PCC的一些中间值,通过阴性或阳性对照来进行对比证明共定位分析的可靠性。
Costes等人提出了一种方法来对任何图像共现或相关性度量进行统计显著性检验(Costes et al., 2004)。为此,对一幅图像进行空间“加扰”——将像素值重新分配给图像内的随机位置。一旦对图像其中一个通道加扰,就可以与另一个通道图像(保持不变)进行比较,并计算出共定位分析的定量值如MOC。然后重复加扰和重新计算定量值。然后将生成的随机值集合与原始图像之间的“真实”值进行比较。通常,如果真正的定量分析值超过了加扰值的95%,它可以被认为具有统计学意义的。该方法虽然稳健,但需要大量的运算过程。
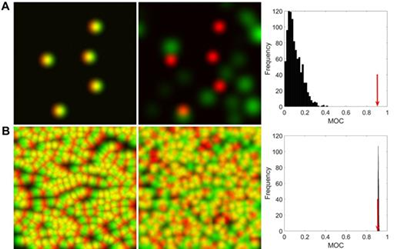
通过图9可以发现,虽然MOC值都是0.90左右,但是B图通过随机显著性分析后排除了共定位的可能,也再次说明MOC作为共定位分析参数的不可靠性。
Dunn等人提出的另一种方法,可以通过相对于一个通道旋转另一个通道的图像、相对于一个通道移动另一个通道的图像或选择两个图像的不同区域进行显著性分析。这种情况本质上与两个实验组的显著性分析相同,不同之处在于,这种方法将一组实验组的平均值与一组错配的数据的平均值进行统计比较。
5. 结语
需要注意的是,图像相关性和共生性都不是分子相互作用的直接测量。传统显微镜的分辨能力被限制在发射光波长的大约一半,而生物分子之间的典型相互作用距离为<10nm。即使随着超分辨率成像技术的出现,分子内相互作用也无法明确观察到。只有近场生物物理技术,如Förster共振能量转移(FRET),才能用于直接测量分子相互作用。然而,相关性和共现性提供了一种推断这种关系的方法。
参考文献
Jesse S. Aaron, Aaron B. Taylor, Teng-Leong Chew; Image co-localization – co-occurrence versus correlation. J Cell Sci 1 February 2018; 131 (3): jcs211847. doi: https://doi.org/10.1242/jcs.211847
Dunn KW, Kamocka MM, McDonald JH. A practical guide to evaluating colocalization in biological microscopy. Am J Physiol Cell Physiol. 2011 Apr;300(4):C723-42. Epub 2011 Jan 5. PMID: 21209361; PMCID: PMC3074624. doi: 10.1152/ajpcell.00462.2010.
Adler, J. and Parmryd, I. (2010), Quantifying colocalization by correlation: The Pearson correlation coefficient is superior to the Mander's overlap coefficient. Cytometry, 77A: 733-742. https://doi.org/10.1002/cyto.a.20896
Costes, S.V., Daelemans, D., Cho, E.H., Dobbin, Z., Pavlakis, G., Lockett, S., 2004. Automatic and Quantitative Measurement of Protein-Protein Colocalization in Live Cells. Biophysical Journal 86, 3993–4003. https://doi.org/10.1529/biophysj.103.038422
